Xây dựng một hệ thống Agentic RAG có khả năng tự cải tiến

Các agent chuyên biệt, đánh giá đa chiều, mặt trận Pareto và hơn thế nữa.
Các hệ thống Agentic RAG hoạt động như một không gian vector đa chiều, trong đó mỗi chiều đại diện cho một quyết định thiết kế như prompt engineering, phối hợp agent, chiến lược truy xuất, và nhiều hơn nữa. Việc tinh chỉnh thủ công các chiều này để tìm ra sự kết hợp đúng đắn là vô cùng khó khăn và dữ liệu chưa từng thấy trong môi trường production thường phá vỡ những gì đã hoạt động tốt trong quá trình thử nghiệm.
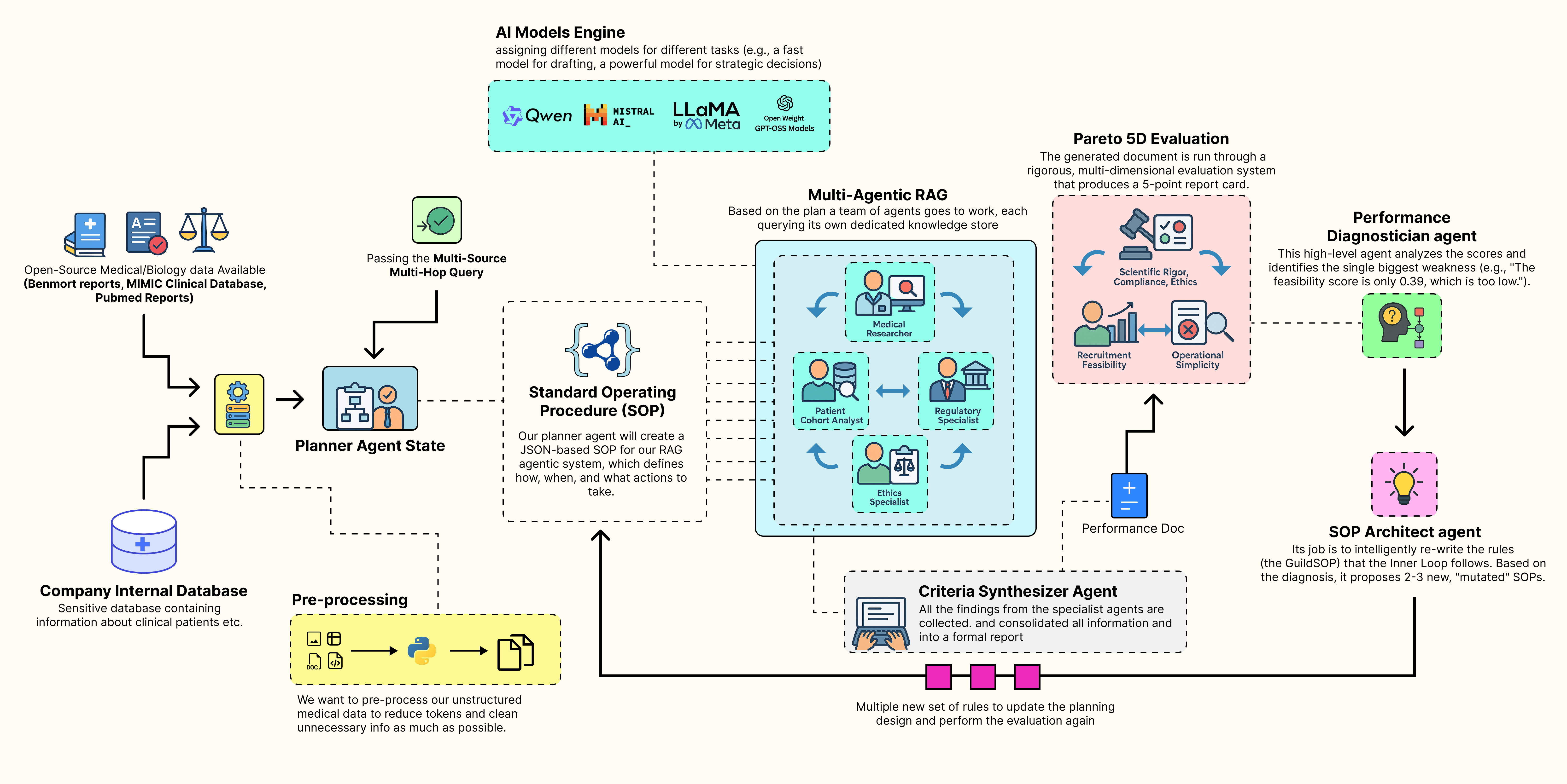
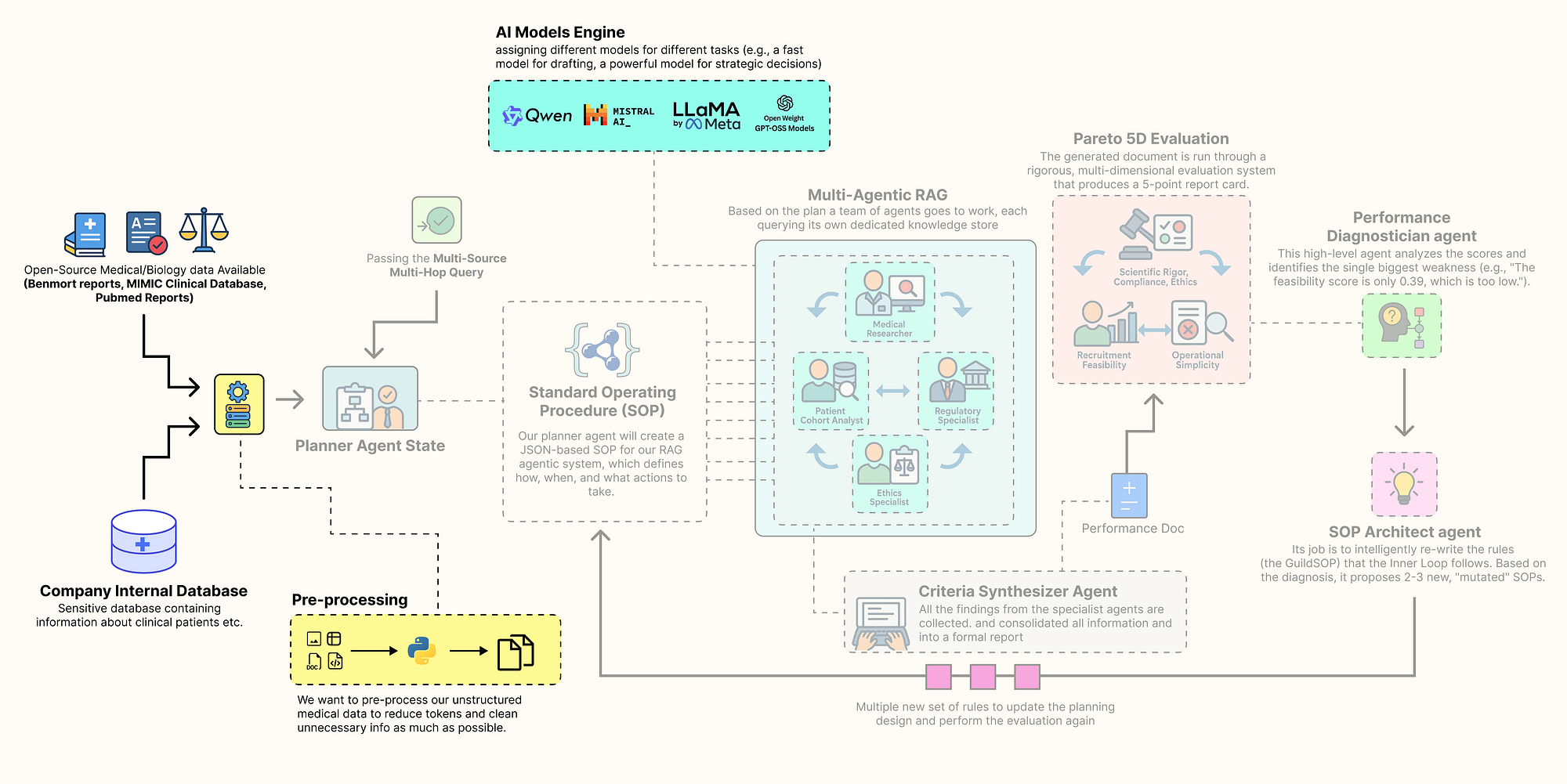
Một cách tiếp cận tốt hơn là để hệ thống tự học cách tối ưu hóa chính nó. Một pipeline Agentic RAG điển hình có khả năng tự phát triển sẽ tuân theo quy trình tư duy như hình dưới đây:
 Hệ thống Agentic RAG tự cải tiến (Tạo bởi Fareed Khan)
Hệ thống Agentic RAG tự cải tiến (Tạo bởi Fareed Khan)
- Một nhóm cộng tác gồm các agent chuyên biệt thực hiện nhiệm vụ. Nó lấy một khái niệm cấp cao và tạo ra một tài liệu hoàn chỉnh, đa nguồn bằng cách sử dụng các quy trình vận hành tiêu chuẩn hiện tại của nó.
- Một hệ thống đánh giá đa chiều chấm điểm đầu ra của nhóm, đo lường hiệu suất trên nhiều mục tiêu như độ chính xác, tính khả thi và sự tuân thủ, tạo ra một vector hiệu suất.
- Một agent chẩn đoán hiệu suất phân tích vector này, hoạt động như một nhà tư vấn để xác định điểm yếu chính trong quy trình và truy tìm nguyên nhân gốc rễ của nó.
- Một agent kiến trúc sư SOP (Quy trình Vận hành Tiêu chuẩn) sử dụng thông tin này để cập nhật các quy trình, đề xuất các biến thể mới được thiết kế đặc biệt để khắc phục điểm yếu đã xác định.
- Mỗi phiên bản mới của SOP được kiểm tra khi nhóm lặp lại nhiệm vụ, với mỗi đầu ra được đánh giá lại để tạo ra vector hiệu suất riêng.
- Hệ thống xác định mặt trận Pareto, là những đánh đổi tốt nhất trong số tất cả các SOP đã được kiểm tra và trình bày các chiến lược tối ưu này cho một người ra quyết định, hoàn thành vòng lặp tiến hóa.
Trong blog này, chúng ta sẽ nhắm đến lĩnh vực chăm sóc sức khỏe, một lĩnh vực rất thách thức vì nhiều khả năng cần được xem xét dựa trên truy vấn đầu vào hoặc cơ sở tri thức, trong khi quyết định cuối cùng vẫn nằm trong tay con người.
Chúng ta sẽ xây dựng một pipeline Agentic RAG tự cải tiến hoàn chỉnh từ đầu đến cuối, tạo ra các mẫu thiết kế khác nhau cho hệ thống RAG.
Toàn bộ mã nguồn có sẵn trong kho GitHub của tôi:
GitHub - FareedKhan-dev/autonomous-agentic-rag: Self improving agentic rag pipeline
Self improving agentic rag pipeline. Contribute to FareedKhan-dev/autonomous-agentic-rag development by creating an…
github.com
Mục lục
- Cơ sở hạ tầng tri thức cho AI y tế ∘ Cài đặt bộ công cụ mã nguồn mở ∘ Cấu hình môi trường & Import ∘ Cấu hình LLM cục bộ ∘ Chuẩn bị các kho tri thức
- Xây dựng mạng lưới thiết kế thử nghiệm nội bộ ∘ Định nghĩa SOP của Guild ∘ Định nghĩa các agent chuyên biệt ∘ Điều phối Guild với LangGraph ∘ Chạy thử toàn bộ Guild Graph
- Hệ thống đánh giá đa chiều ∘ Xây dựng một trình đánh giá tùy chỉnh cho mỗi tham số ∘ Tạo trình đánh giá tổng hợp LangSmith
- Vòng lặp ngoài của bộ máy tiến hóa ∘ Quản lý cấu hình Guild ∘ Xây dựng các agent cấp giám đốc ∘ Chạy toàn bộ vòng lặp tiến hóa
- Phân tích dựa trên Pareto 5D ∘ Xác định mặt trận Pareto ∘ Trực quan hóa mặt trận & Ra quyết định
- Hiểu luồng công việc nhận thức ∘ Trực quan hóa dòng thời gian của luồng công việc agent ∘ Phân tích đầu ra bằng biểu đồ radar
- Biến nó thành một chiến lược tự trị
Cơ sở hạ tầng tri thức cho AI y tế
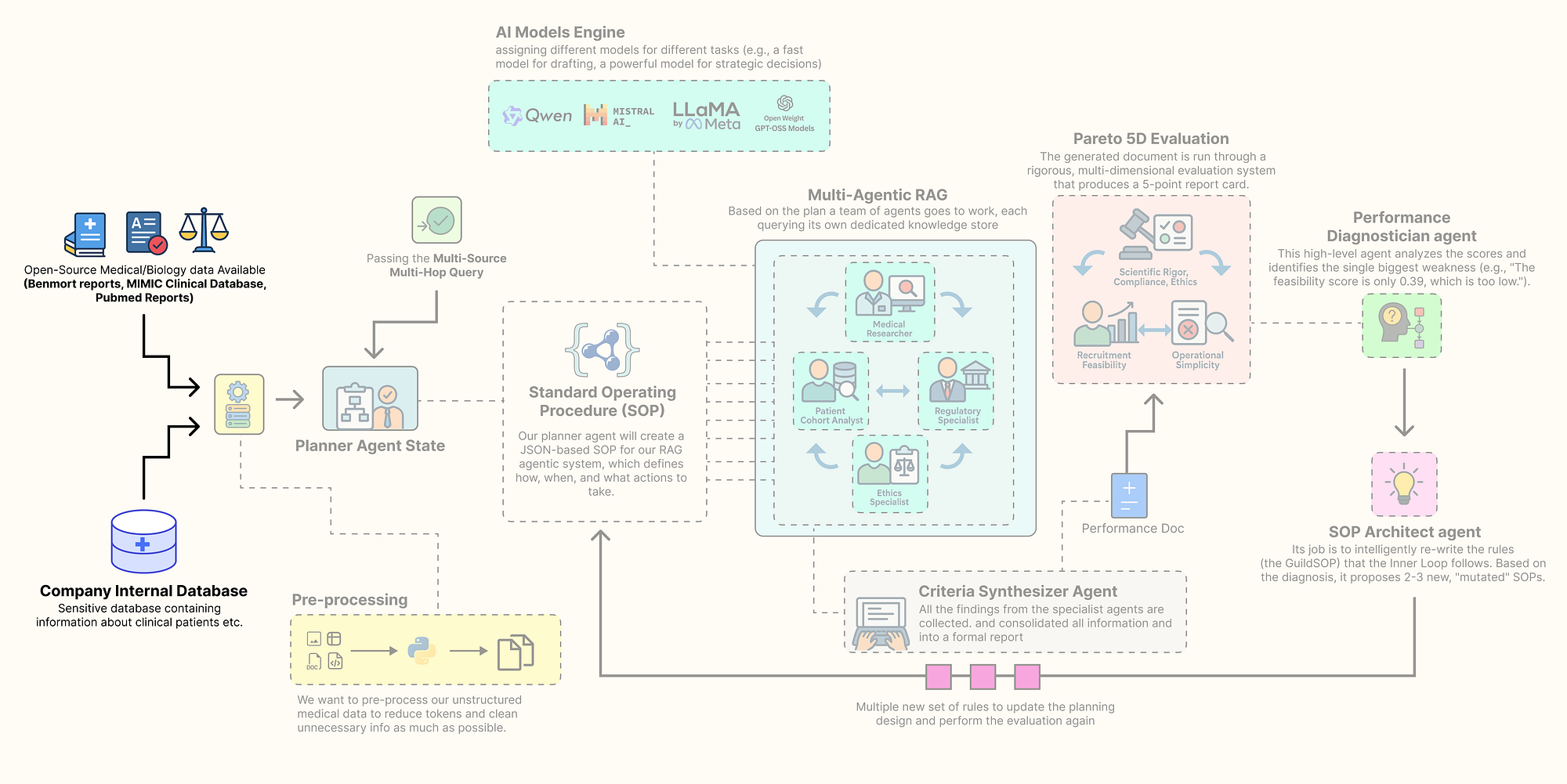
Trước khi có thể lập trình hệ thống Agentic RAG tự tiến hóa, chúng ta cần thiết lập một cơ sở dữ liệu tri thức phù hợp cùng với các công cụ cần thiết để xây dựng kiến trúc.
Một hệ thống RAG cấp production thường chứa một tập hợp đa dạng các cơ sở dữ liệu, bao gồm dữ liệu nhạy cảm của tổ chức cũng như dữ liệu mã nguồn mở, để cải thiện chất lượng truy xuất và bù đắp cho thông tin lỗi thời hoặc không đầy đủ. Bước nền tảng này được cho là quan trọng nhất…
vì chất lượng của các nguồn dữ liệu sẽ quyết định trực tiếp chất lượng của đầu ra cuối cùng của chúng ta.
 Tìm nguồn cung cấp cơ sở tri thức (Tạo bởi Fareed Khan)
Tìm nguồn cung cấp cơ sở tri thức (Tạo bởi Fareed Khan)
Trong phần này, chúng ta sẽ lắp ráp mọi thành phần của kiến trúc này. Đây là những gì chúng ta sẽ làm:
- Cài đặt bộ công cụ mã nguồn mở: Chúng ta sẽ thiết lập môi trường với tất cả các thư viện cần thiết, tập trung vào cách tiếp cận ưu tiên mã nguồn mở và cục bộ.
- Cấu hình khả năng quan sát an toàn: Sau đó, chúng ta sẽ tải các khóa API một cách an toàn và cấu hình
LangSmithđể theo dõi và gỡ lỗi các tương tác agent phức tạp của chúng ta ngay từ đầu. - Xây dựng một lò đúc LLM cục bộ: Chúng ta sẽ xây dựng một bộ các mô hình mã nguồn mở khác nhau bằng
Ollama, gán các mô hình cụ thể cho các nhiệm vụ cụ thể để tối ưu hóa hiệu suất và chi phí. - Tìm nguồn và xử lý dữ liệu đa phương thức: tải xuống và chuẩn bị bốn nguồn dữ liệu thực tế: tài liệu khoa học từ PubMed, hướng dẫn quy định từ FDA, các nguyên tắc đạo đức, và một bộ dữ liệu lâm sàng có cấu trúc khổng lồ (MIMIC-III).
- Lập chỉ mục các kho tri thức: Cuối cùng, chúng ta sẽ xử lý dữ liệu thô này thành các cơ sở dữ liệu có khả năng tìm kiếm hiệu quả cao, các kho vector
FAISScho văn bản phi cấu trúc và một instanceDuckDBcho dữ liệu lâm sàng có cấu trúc của chúng ta.
Cài đặt bộ công cụ mã nguồn mở
Vậy, bước đầu tiên của chúng ta là cài đặt tất cả các thư viện Python cần thiết. Một môi trường có thể tái tạo là nền tảng của bất kỳ dự án nghiêm túc nào. Chúng ta đang chọn một bộ công cụ mã nguồn mở, tiêu chuẩn ngành, cho phép chúng ta kiểm soát hoàn toàn hệ thống của mình. Điều này bao gồm langchain và langgraph cho framework agent cốt lõi, ollama để tương tác với các LLM cục bộ của chúng ta, và các thư viện chuyên dụng như biopython để truy cập PubMed và duckdb cho phân tích hiệu suất cao trên dữ liệu lâm sàng của chúng ta.
Hãy cài đặt các module cần thiết…
# Chúng ta sử dụng cờ "quiet" (-q) và "upgrade" (-U) của pip để cài đặt tất cả các gói cần thiết.
# - langchain, langgraph, v.v.: Đây là những thành phần cốt lõi của framework agent để xây dựng và điều phối các agent.
# - ollama: Đây là thư viện client cho phép mã Python của chúng ta giao tiếp với một máy chủ Ollama đang chạy cục bộ.
# - duckdb: Một cơ sở dữ liệu phân tích trong tiến trình cực kỳ nhanh, hoàn hảo để xử lý dữ liệu MIMIC có cấu trúc của chúng ta mà không cần một thiết lập máy chủ nặng nề.
# - faiss-cpu: Thư viện của Facebook AI cho tìm kiếm tương đồng hiệu quả, sẽ cung cấp năng lượng cho các kho vector của các agent RAG của chúng ta.
# - sentence-transformers: Một thư viện để dễ dàng truy cập các mô hình tiên tiến nhất để tạo các embedding văn bản.
# - biopython, pypdf, beautifulsoup4: Một bộ các tiện ích mạnh mẽ để tải xuống và phân tích cú pháp các nguồn dữ liệu đa dạng, thực tế của chúng ta.
%pip install -U langchain langgraph langchain_community langchain_openai langchain_core ollama pandas duckdb faiss-cpu sentence-transformers biopython pypdf pydantic lxml html2text beautifulsoup4 matplotlib -qqq
Chúng ta đang thu thập tất cả các công cụ và vật liệu xây dựng mà chúng ta sẽ cần cho phần còn lại của dự án trong một lần. Mỗi thư viện có một vai trò cụ thể, từ các luồng công việc của agent với langgraph đến phân tích dữ liệu với duckdb.
Bây giờ chúng ta đã cài đặt các module cần thiết, hãy bắt đầu khởi tạo chúng từng cái một.
Cấu hình môi trường & Import
Chúng ta cần cấu hình môi trường một cách an toàn. Việc mã hóa cứng các khóa API trực tiếp vào notebook là một rủi ro bảo mật đáng kể và làm cho mã khó chia sẻ.
Chúng ta sẽ sử dụng tệp .env để quản lý các bí mật của mình, chủ yếu là khóa API LangSmith. Việc thiết lập LangSmith ngay từ đầu là không thể thiếu đối với một dự án có độ phức tạp như thế này, nó cung cấp khả năng quan sát sâu mà chúng ta sẽ cần để theo dõi, gỡ lỗi và hiểu các tương tác giữa các agent của mình. Vậy, hãy làm điều đó.
import os
import getpass
from dotenv import load_dotenv
# Hàm này từ thư viện python-dotenv tìm kiếm tệp .env và tải các cặp khóa-giá trị của nó
# vào các biến môi trường của hệ điều hành, làm cho chúng có thể truy cập được bởi kịch bản của chúng ta.
load_dotenv()
# Đây là một kiểm tra quan trọng. Chúng ta xác minh rằng kịch bản của chúng ta có thể truy cập các khóa API cần thiết từ môi trường.
if "LANGCHAIN_API_KEY" not in os.environ or "ENTREZ_EMAIL" not in os.environ:
# Nếu các khóa bị thiếu, chúng ta in ra một lỗi và dừng lại, vì ứng dụng không thể tiếp tục.
print("Các biến môi trường bắt buộc chưa được thiết lập. Vui lòng thiết lập chúng trong tệp .env hoặc môi trường của bạn.")
else:
# Xác nhận này cho chúng ta biết các bí mật của chúng ta đã được tải một cách an toàn và sẵn sàng để sử dụng.
print("Các biến môi trường đã được tải thành công.")
# Chúng ta đặt tên dự án LangSmith một cách tường minh. Đây là một thực hành tốt nhất đảm bảo tất cả các dấu vết
# được tạo ra bởi dự án này sẽ tự động được nhóm lại với nhau trong giao diện người dùng LangSmith để dễ dàng phân tích.
os.environ["LANGCHAIN_PROJECT"] = "AI_Clinical_Trials_Architect"
Hàm load_dotenv() hoạt động như một cầu nối an toàn giữa các thông tin nhạy cảm và mã của chúng ta. Nó đọc tệp .env (không bao giờ nên được commit vào hệ thống quản lý phiên bản) và đưa các khóa vào môi trường phiên làm việc của chúng ta.
Từ thời điểm này trở đi, mọi hoạt động chúng ta thực hiện với LangChain hoặc LangGraph sẽ được tự động ghi lại và gửi đến dự án của chúng ta trong LangSmith.
Cấu hình LLM cục bộ
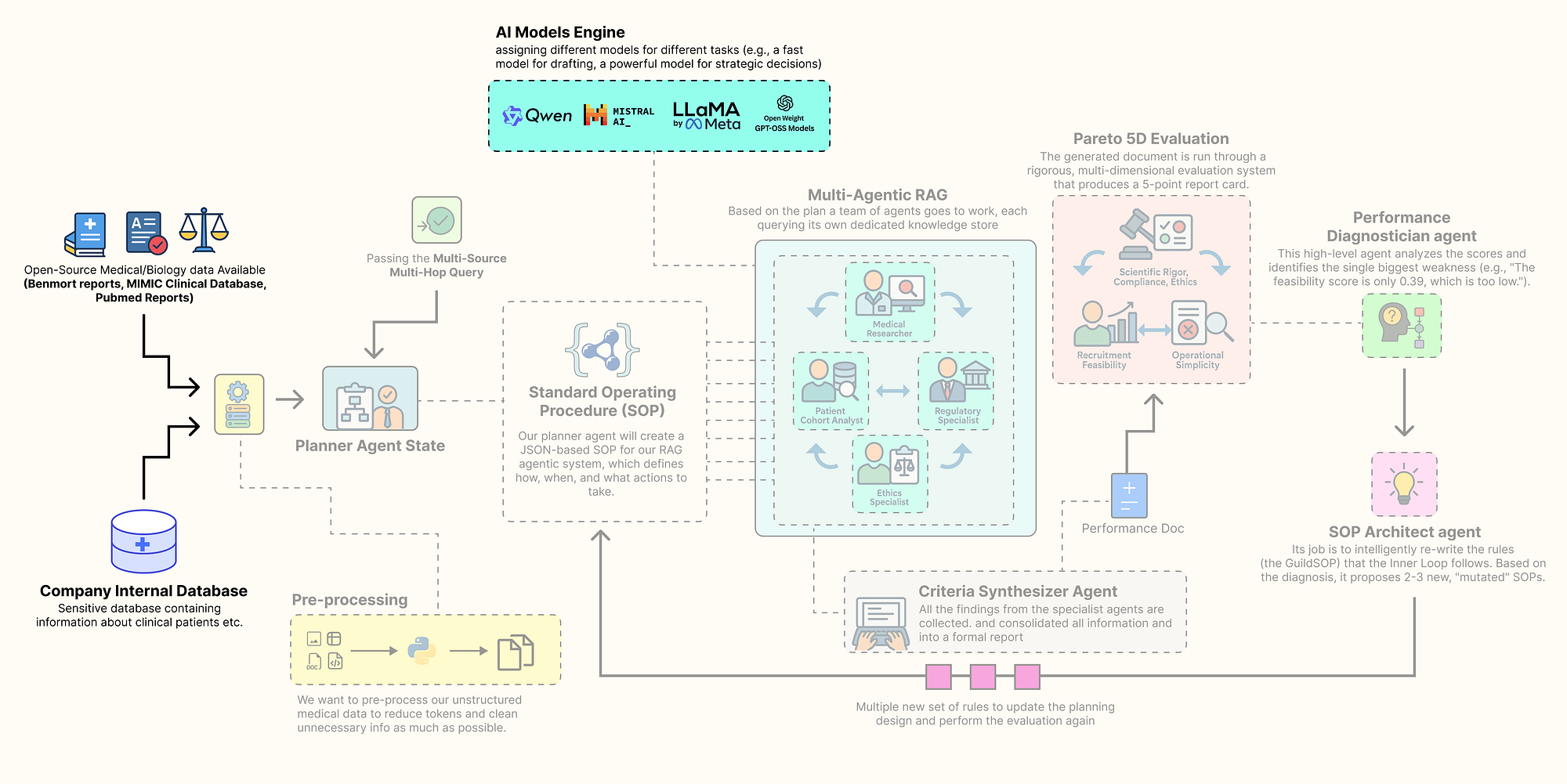
Trong các hệ thống agent cấp production, chiến lược một mô hình cho tất cả hiếm khi là tối ưu. Một mô hình lớn, tiên tiến nhất rất tốn kém về mặt tính toán và chậm chạp, việc sử dụng nó cho mọi tác vụ đơn giản sẽ là lãng phí tài nguyên, đặc biệt nếu nó được lưu trữ trên GPU của bạn. Nhưng một mô hình nhỏ, nhanh có thể thiếu sức mạnh suy luận sâu cần thiết cho các quyết định chiến lược có tính rủi ro cao.
 Cấu hình LLM cục bộ (Tạo bởi Fareed Khan)
Cấu hình LLM cục bộ (Tạo bởi Fareed Khan)
Chìa khóa là đặt đúng mô hình vào đúng vị trí trong hệ thống agent của bạn. Chúng ta sẽ xây dựng một nhóm các mô hình mã nguồn mở khác nhau, mỗi mô hình được chọn vì thế mạnh của nó trong một vai trò cụ thể, và tất cả đều được phục vụ cục bộ qua Ollama để đảm bảo quyền riêng tư, kiểm soát và hiệu quả chi phí.
Chúng ta cần định nghĩa một từ điển cấu hình để chứa các client cho mỗi mô hình đã chọn. Bằng cách này, chúng ta có thể dễ dàng hoán đổi các mô hình và tập trung hóa việc quản lý mô hình của mình.
from langchain_community.chat_models import ChatOllama
from langchain_community.embeddings import OllamaEmbeddings
# Từ điển này sẽ hoạt động như một sổ đăng ký trung tâm, hay "lò đúc," cho tất cả các client mô hình LLM và embedding.
llm_config = {
# Đối với 'planner', chúng ta sử dụng Llama 3.1 8B. Đây là một mô hình hiện đại, có khả năng cao, xuất sắc trong việc tuân thủ chỉ dẫn.
# Chúng ta đặt `format='json'` để tận dụng chế độ JSON tích hợp của Ollama, đảm bảo đầu ra có cấu trúc đáng tin cậy cho nhiệm vụ quan trọng này.
"planner": ChatOllama(model="llama3.1:8b-instruct", temperature=0.0, format='json'),
# Đối với 'drafter' và 'sql_coder', chúng ta sử dụng Qwen2 7B. Đây là một mô hình nhanh nhẹn và tốc độ, hoàn hảo cho
# các tác vụ như tạo văn bản và hoàn thành mã nơi tốc độ là có giá trị.
"drafter": ChatOllama(model="qwen2:7b", temperature=0.2),
"sql_coder": ChatOllama(model="qwen2:7b", temperature=0.0),
# Đối với 'director', agent chiến lược cấp cao nhất, chúng ta sử dụng mô hình Llama 3 70B mạnh mẽ.
# Nhiệm vụ có tính rủi ro cao này là chẩn đoán hiệu suất và phát triển các quy trình của chính hệ thống
# biện minh cho việc sử dụng một mô hình lớn hơn, mạnh mẽ hơn.
"director": ChatOllama(model="llama3:70b", temperature=0.0, format='json'),
# Đối với embeddings, chúng ta sử dụng 'nomic-embed-text', một mô hình mã nguồn mở hiệu quả, hàng đầu.
"embedding_model": OllamaEmbeddings(model="nomic-embed-text")
}
Vậy là chúng ta vừa tạo ra từ điển llm_config, đóng vai trò là một trung tâm tập trung cho tất cả các khởi tạo mô hình của chúng ta. Bằng cách gán các mô hình khác nhau cho các vai trò khác nhau, chúng ta đang tạo ra một hệ thống phân cấp được tối ưu hóa về chi phí và hiệu suất.
- Nhanh & Nhẹ (mô hình 7B-8B): Các vai trò
planner,drafter, vàsql_coderxử lý các tác vụ thường xuyên, được xác định rõ ràng. Sử dụng các mô hình nhỏ hơn nhưQwen2 7BvàLlama 3.1 8Bcho các vai trò này đảm bảo độ trễ thấp và sử dụng tài nguyên hiệu quả. Chúng hoàn toàn có khả năng tuân theo chỉ dẫn để tạo kế hoạch, soạn thảo văn bản, hoặc viết SQL. - Sâu & Chiến lược (mô hình 70B): Agent
directorcó công việc phức tạp nhất, nó phải phân tích dữ liệu hiệu suất đa chiều và viết lại toàn bộ quy trình vận hành của hệ thống. Điều này đòi hỏi suy luận sâu và sự hiểu biết về nguyên nhân và kết quả. Đối với nhiệm vụ có tính rủi ro cao, tần suất thấp này, chúng ta phân bổ tài nguyên mạnh nhất của mình, mô hìnhLlama 3 70B.
Hãy thực thi ô này để khởi tạo các client và in ra cấu hình của chúng.
# In cấu hình để xác nhận các client đã được khởi tạo và các tham số của chúng được đặt chính xác.
print("Các client LLM đã được cấu hình:")
print(f"Planner ({llm_config['planner'].model}): {llm_config['planner']}")
print(f"Drafter ({llm_config['drafter'].model}): {llm_config['drafter']}")
print(f"SQL Coder ({llm_config['sql_coder'].model}): {llm_config['sql_coder']}")
print(f"Director ({llm_config['director'].model}): {llm_config['director']}")
print(f"Embedding Model ({llm_config['embedding_model'].model}): {llm_config['embedding_model']}")
Đây là những gì chúng ta nhận được…
OUTPUT
LLM clients configured:
Planner (llama3.1:8b-instruct): ChatOllama(model='llama3.1:8b-instruct', temperature=0.0, format='json')
Drafter (qwen2:7b): ChatOllama(model='qwen2:7b', temperature=0.2)
SQL Coder (qwen2:7b): ChatOllama(model='qwen2:7b', temperature=0.0)
Director (llama3:70b): ChatOllama(model='llama3:70b', temperature=0.0, format='json')
Embedding Model (nomic-embed-text): OllamaEmbeddings(model='nomic-embed-text')
Đầu ra xác nhận rằng các client ChatOllama và OllamaEmbeddings của chúng ta đã được khởi tạo thành công với các mô hình và tham số tương ứng. Bây giờ chúng ta đã sẵn sàng để kết nối với các kho tri thức của mình.
Chuẩn bị các kho tri thức
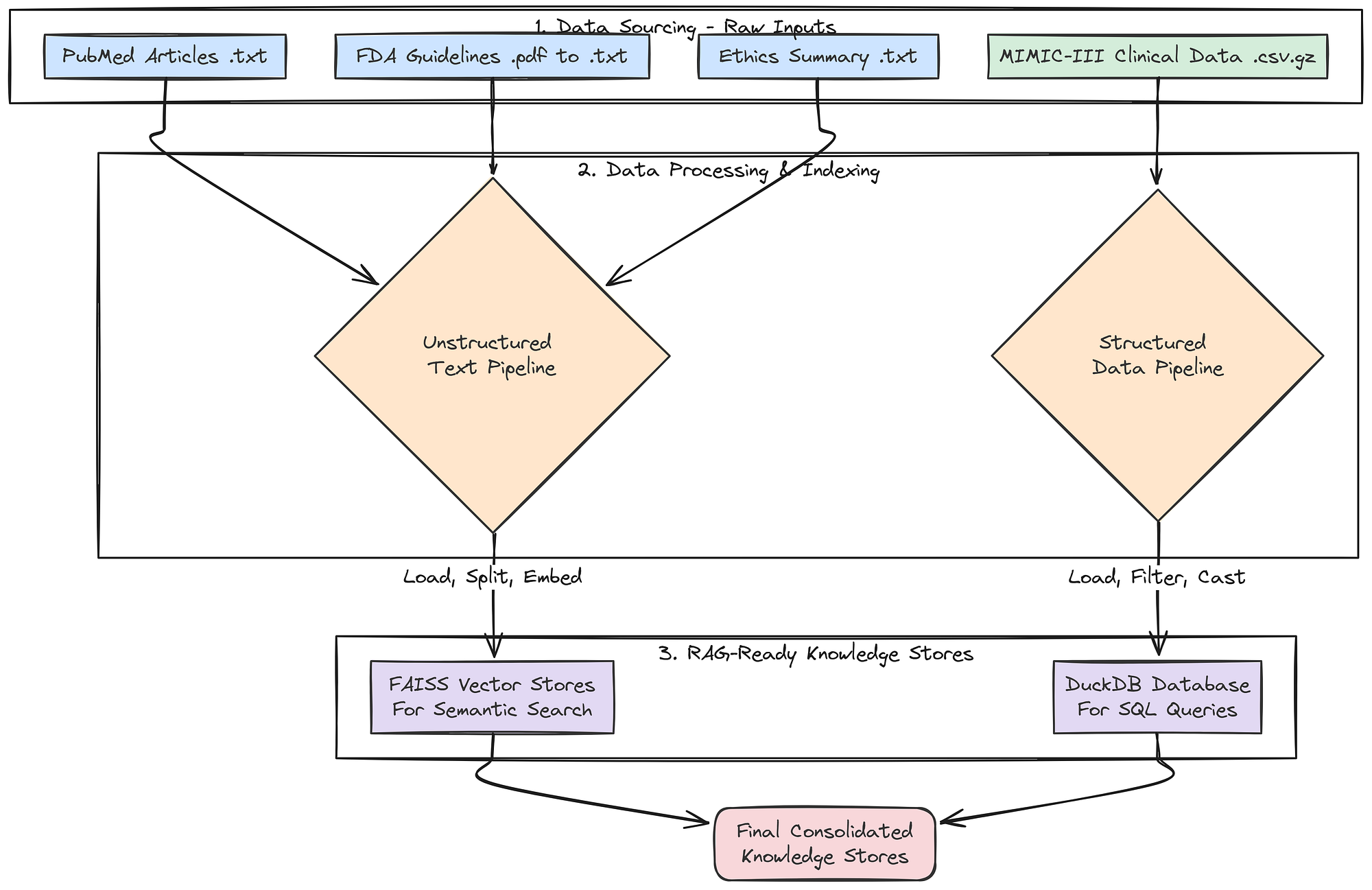
Phần quan trọng nhất của RAG là đây, một cơ sở tri thức đa phương thức phong phú để dựa vào. Một tìm kiếm dựa trên web chung chung là không đủ cho một nhiệm vụ chuyên biệt như thiết kế thử nghiệm lâm sàng. Chúng ta cần đặt các agent của mình vào thông tin có thẩm quyền, chuyên ngành.
 Tạo kho tri thức (Tạo bởi Fareed Khan)
Tạo kho tri thức (Tạo bởi Fareed Khan)
Để đạt được điều này, bây giờ chúng ta sẽ xây dựng một cơ sở tri thức toàn diện bằng cách tìm nguồn, tải xuống và xử lý bốn loại dữ liệu thực tế khác nhau. Cách tiếp cận đa nguồn này rất quan trọng để cho phép các agent của chúng ta tổng hợp thông tin và tạo ra một đầu ra toàn diện, đầy đủ.
Đầu tiên, một bước nhỏ nhưng quan trọng: chúng ta sẽ tạo các thư mục nơi dữ liệu đã tải xuống và xử lý của chúng ta sẽ được lưu trữ.
import os
# Một từ điển để chứa các đường dẫn cho các loại dữ liệu khác nhau của chúng ta. Điều này giữ cho việc quản lý tệp của chúng ta sạch sẽ và tập trung.
data_paths = {
"base": "./data",
"pubmed": "./data/pubmed_articles",
"fda": "./data/fda_guidelines",
"ethics": "./data/ethical_guidelines",
"mimic": "./data/mimic_db"
}
# Vòng lặp này lặp qua các đường dẫn đã định nghĩa của chúng ta và sử dụng os.makedirs() để tạo bất kỳ thư mục nào chưa tồn tại.
# Điều này ngăn ngừa lỗi trong các bước sau khi chúng ta cố gắng lưu tệp vào các vị trí này.
for path in data_paths.values():
if not os.path.exists(path):
os.makedirs(path)
print(f"Đã tạo thư mục: {path}")
Chúng ta đang đảm bảo dự án của mình có một cấu trúc tệp sạch sẽ và có tổ chức ngay từ đầu. Bằng cách định nghĩa trước và tạo các thư mục này, các hàm xử lý dữ liệu tiếp theo của chúng ta trở nên mạnh mẽ hơn, chúng có thể lưu đầu ra của mình một cách đáng tin cậy vào đúng vị trí mà không cần kiểm tra xem thư mục có tồn tại trước hay không.
Tiếp theo, chúng ta sẽ lấy tài liệu khoa học thực tế từ PubMed. Điều này sẽ cung cấp kiến thức cốt lõi cho agent Medical Researcher của chúng ta, đặt nền tảng cho công việc của nó trong khoa học được bình duyệt, cập nhật.
from Bio import Entrez
from Bio import Medline
def download_pubmed_articles(query, max_articles=20):
"""Lấy các tóm tắt từ PubMed cho một truy vấn nhất định và lưu chúng dưới dạng tệp văn bản."""
# API của NCBI yêu cầu một địa chỉ email để nhận dạng. Chúng ta lấy nó từ các biến môi trường của mình.
Entrez.email = os.environ.get("ENTREZ_EMAIL")
print(f"Đang lấy các bài báo PubMed cho truy vấn: {query}")
# Bước 1: Sử dụng Entrez.esearch để tìm các ID PubMed (PMID) cho các bài báo khớp với truy vấn của chúng ta.
handle = Entrez.esearch(db="pubmed", term=query, retmax=max_articles, sort="relevance")
record = Entrez.read(handle)
id_list = record["IdList"]
print(f"Tìm thấy {len(id_list)} ID bài báo.")
print("Đang tải xuống các bài báo...")
# Bước 2: Sử dụng Entrez.efetch để lấy các bản ghi đầy đủ (ở định dạng MEDLINE) cho danh sách các PMID.
handle = Entrez.efetch(db="pubmed", id=id_list, rettype="medline", retmode="text")
records = Medline.parse(handle)
count = 0
# Bước 3: Lặp qua các bản ghi đã lấy, phân tích cú pháp chúng và lưu mỗi tóm tắt vào một tệp.
for i, record in enumerate(records):
pmid = record.get("PMID", "")
title = record.get("TI", "No Title")
abstract = record.get("AB", "No Abstract")
if pmid:
# Chúng ta đặt tên tệp theo PMID để dễ tham chiếu và tránh trùng lặp.
filepath = os.path.join(data_paths["pubmed"], f"{pmid}.txt")
with open(filepath, "w") as f:
f.write(f"Title: {title}\n\nAbstract: {abstract}")
print(f"[{i+1}/{len(id_list)}] Đang lấy PMID: {pmid}... Đã lưu vào {filepath}")
count += 1
return count
Hàm download_pubmed_articles là kết nối trực tiếp của chúng ta với tài liệu khoa học trực tuyến. Đó là một quy trình ba bước:
- Tìm kiếm các ID bài báo có liên quan.
- Lấy các bản ghi đầy đủ cho các ID đó.
- Phân tích và lưu các tóm tắt vào các tệp văn bản.
Hãy chạy hàm này với một truy vấn cụ thể cho trường hợp sử dụng của chúng ta.
# Chúng ta định nghĩa một truy vấn boolean cụ thể để tìm các bài báo có liên quan cao đến khái niệm thử nghiệm của chúng ta.
pubmed_query = "(SGLT2 inhibitor) AND (type 2 diabetes) AND (renal impairment)"
num_downloaded = download_pubmed_articles(pubmed_query)
print(f"Tải xuống PubMed hoàn tất. Đã lưu {num_downloaded} bài báo.")
Khi chúng ta chạy mã trên, nó sẽ bắt đầu tải xuống các bài báo pubmed có liên quan cao đến truy vấn của chúng ta.
OUTPUT
Fetching PubMed articles for query: (SGLT2 inhibitor) AND (type 2 diabetes) AND (renal impairment)
Found 20 article IDs.
Downloading articles...
[1/20] Fetching PMID: 38810260... Saved to ./data/pubmed_articles/38810260.txt
[2/20] Fetching PMID: 38788484... Saved to ./data/pubmed_articles/38788484.txt
...
PubMed download complete. 20 articles saved.
Nó đã kết nối thành công với cơ sở dữ liệu NCBI, thực hiện truy vấn cụ thể của chúng ta và tải xuống 20 tóm tắt khoa học có liên quan, lưu mỗi cái vào thư mục pubmed_articles được chỉ định của chúng ta.
Agent Medical Researcher của chúng ta bây giờ sẽ có một cơ sở tri thức phong phú, hiện tại và chuyên ngành để tham khảo, đảm bảo các phát hiện của nó được đặt nền tảng trên khoa học thực tế.
Bây giờ, hãy lấy các tài liệu quy định mà agent Regulatory Specialist của chúng ta sẽ cần. Một phần quan trọng của thiết kế thử nghiệm là đảm bảo tuân thủ các hướng dẫn của chính phủ.
import requests
from pypdf import PdfReader
import io
def download_and_extract_text_from_pdf(url, output_path):
"""Tải xuống một tệp PDF từ một URL, lưu nó, và cũng trích xuất nội dung văn bản của nó vào một tệp .txt riêng biệt."""
print(f"Đang tải xuống Hướng dẫn của FDA: {url}")
try:
# Chúng ta sử dụng thư viện 'requests' để thực hiện yêu cầu HTTP GET để tải xuống tệp.
response = requests.get(url)
response.raise_for_status() # Đây là một thực hành tốt sẽ gây ra lỗi nếu việc tải xuống thất bại (ví dụ: lỗi 404).
# Chúng ta lưu tệp PDF thô, hữu ích cho mục đích lưu trữ.
with open(output_path, 'wb') as f:
f.write(response.content)
print(f"Đã tải xuống và lưu thành công vào {output_path}")
# Sau đó, chúng ta sử dụng pypdf để đọc nội dung PDF trực tiếp từ phản hồi trong bộ nhớ.
reader = PdfReader(io.BytesIO(response.content))
text = ""
# Chúng ta lặp qua từng trang của PDF và nối văn bản đã trích xuất của nó.
for page in reader.pages:
text += page.extract_text() + "\n\n"
# Cuối cùng, chúng ta lưu văn bản đã trích xuất, sạch sẽ vào một tệp .txt. Đây là tệp mà hệ thống RAG của chúng ta sẽ thực sự sử dụng.
txt_output_path = os.path.splitext(output_path)[0] + '.txt'
with open(txt_output_path, 'w') as f:
f.write(text)
return True
except requests.exceptions.RequestException as e:
print(f"Lỗi khi tải xuống tệp: {e}")
return False
Hàm này, download_and_extract_text_from_pdf, là công cụ của chúng ta để xử lý các tài liệu PDF. Đó là một quy trình hai giai đoạn.
- Tải xuống và lưu tệp PDF gốc.
- Trích xuất nội dung văn bản và lưu nó vào một tệp
.txtriêng biệt, sẵn sàng cho RAG.
Hãy chạy hàm để tải xuống tài liệu hướng dẫn của FDA.
# URL này trỏ đến một tài liệu hướng dẫn thực tế của FDA để phát triển thuốc cho bệnh tiểu đường.
fda_url = "https://www.fda.gov/media/71185/download"
fda_pdf_path = os.path.join(data_paths["fda"], "fda_diabetes_guidance.pdf")
download_and_extract_text_from_pdf(fda_url, fda_pdf_path)
OUTPUT
Downloading FDA Guideline: https://www.fda.gov/media/71185/download
Successfully downloaded and saved to ./data/fda_guidelines/fda_diabetes_guidance.pdf
Bây giờ chúng ta có cả tệp fda_diabetes_guidance.pdf gốc và phiên bản văn bản đã trích xuất của nó trong thư mục fda_guidelines. Agent Regulatory Specialist của chúng ta giờ đã được trang bị văn bản pháp lý và quy định nền tảng của nó.
Tiếp theo, chúng ta sẽ tạo một tài liệu được tuyển chọn cho Ethics Specialist của mình. Mặc dù chúng ta có thể tìm kiếm thông tin này, việc cung cấp một bản tóm tắt ngắn gọn, có thẩm quyền về các nguyên tắc cốt lõi đảm bảo rằng lý luận của agent được đặt nền tảng trên các khái niệm quan trọng nhất.
# Chuỗi nhiều dòng này chứa một bản tóm tắt được tuyển chọn về ba nguyên tắc cốt lõi của Báo cáo Belmont,
# là tài liệu nền tảng cho đạo đức trong nghiên cứu trên người ở Hoa Kỳ.
ethics_content = """
Title: Summary of the Belmont Report Principles for Clinical Research
1. Respect for Persons: This principle requires that individuals be treated as autonomous agents and that persons with diminished autonomy are entitled to protection. This translates to robust informed consent processes. Inclusion/exclusion criteria must not unduly target or coerce vulnerable populations, such as economically disadvantaged individuals, prisoners, or those with severe cognitive impairments, unless the research is directly intended to benefit that population.
2. Beneficence: This principle involves two complementary rules: (1) do not harm and (2) maximize possible benefits and minimize possible harms. The criteria must be designed to select a population that is most likely to benefit and least likely to be harmed by the intervention. The risks to subjects must be reasonable in relation to anticipated benefits.
3. Justice: This principle concerns the fairness of distribution of the burdens and benefits of research. The selection of research subjects must be equitable. Criteria should not be designed to exclude certain groups without a sound scientific or safety-related justification. For example, excluding participants based on race, gender, or socioeconomic status is unjust unless there is a clear rationale related to the drug's mechanism or risk profile.
"""
# Chúng ta định nghĩa đường dẫn nơi tài liệu đạo đức của chúng ta sẽ được lưu.
ethics_path = os.path.join(data_paths["ethics"], "belmont_summary.txt")
# Chúng ta mở tệp ở chế độ ghi và lưu nội dung.
with open(ethics_path, "w") as f:
f.write(ethics_content)
print(f"Đã tạo tệp hướng dẫn đạo đức: {ethics_path}")
Chúng ta đã tạo một tài liệu tập trung cho Ethics Specialist của mình. Thay vì để agent sàng lọc toàn bộ Báo cáo Belmont, chúng ta đã cung cấp cho nó thông tin quan trọng nhất ở định dạng sạch sẽ, dễ tiêu hóa. Điều này đảm bảo phân tích của nó sẽ nhất quán và được đặt nền tảng trên các nguyên tắc cốt lõi.
Bây giờ đến nguồn dữ liệu phức tạp nhất của chúng ta: dữ liệu lâm sàng có cấu trúc từ MIMIC-III. Điều này sẽ cung cấp dữ liệu dân số thực tế mà Patient Cohort Analyst của chúng ta cần để đánh giá tính khả thi của việc tuyển dụng.
import duckdb
import pandas as pd
import os
def load_real_mimic_data():
"""Tải các tệp CSV MIMIC-III thực vào một tệp cơ sở dữ liệu DuckDB bền vững, xử lý bảng LABEVENTS khổng lồ một cách hiệu quả."""
print("Đang cố gắng tải dữ liệu MIMIC-III thực từ các tệp CSV cục bộ...")
db_path = os.path.join(data_paths["mimic"], "mimic3_real.db")
csv_dir = os.path.join(data_paths["mimic"], "mimiciii_csvs")
# Định nghĩa các đường dẫn đến các tệp CSV nén cần thiết.
required_files = {
"patients": os.path.join(csv_dir, "PATIENTS.csv.gz"),
"diagnoses": os.path.join(csv_dir, "DIAGNOSES_ICD.csv.gz"),
"labevents": os.path.join(csv_dir, "LABEVENTS.csv.gz"),
}
# Trước khi bắt đầu, chúng ta kiểm tra xem tất cả các tệp nguồn cần thiết có tồn tại không.
missing_files = [path for path in required_files.values() if not os.path.exists(path)]
if missing_files:
print("LỖI: Không tìm thấy các tệp MIMIC-III sau:")
for f in missing_files: print(f"- {f}")
print("\nVui lòng tải chúng xuống theo hướng dẫn và đặt chúng vào thư mục chính xác.")
return None
print("Đã tìm thấy các tệp cần thiết. Tiếp tục tạo cơ sở dữ liệu.")
# Xóa bất kỳ tệp cơ sở dữ liệu cũ nào để đảm bảo chúng ta đang xây dựng từ đầu.
if os.path.exists(db_path):
os.remove(db_path)
# Kết nối với DuckDB. Nếu tệp cơ sở dữ liệu không tồn tại, nó sẽ được tạo.
con = duckdb.connect(db_path)
# Sử dụng `read_csv_auto` mạnh mẽ của DuckDB để tải trực tiếp dữ liệu từ các tệp CSV gzipped vào các bảng SQL.
print(f"Đang tải {required_files['patients']} vào DuckDB...")
con.execute(f"CREATE TABLE patients AS SELECT SUBJECT_ID, GENDER, DOB, DOD FROM read_csv_auto('{required_files['patients']}')")
print(f"Đang tải {required_files['diagnoses']} vào DuckDB...")
con.execute(f"CREATE TABLE diagnoses_icd AS SELECT SUBJECT_ID, ICD9_CODE FROM read_csv_auto('{required_files['diagnoses']}')")
# Bảng LABEVENTS rất lớn. Để xử lý nó một cách mạnh mẽ, chúng ta sử dụng một quy trình hai giai đoạn.
print(f"Đang tải và xử lý {required_files['labevents']} (quá trình này có thể mất vài phút)...")
# 1. Tải dữ liệu vào một bảng 'staging' tạm thời, coi tất cả các cột là văn bản (`all_varchar=True`).
# Điều này ngăn ngừa lỗi phân tích cú pháp với các loại dữ liệu hỗn hợp. Chúng ta cũng chỉ lọc các ID mục xét nghiệm mà chúng ta
# quan tâm (50912 cho Creatinine, 50852 cho HbA1c) và sử dụng regex để đảm bảo VALUENUM là số.
con.execute(f"""CREATE TABLE labevents_staging AS
SELECT SUBJECT_ID, ITEMID, VALUENUM
FROM read_csv_auto('{required_files['labevents']}', all_varchar=True)
WHERE ITEMID IN ('50912', '50852') AND VALUENUM IS NOT NULL AND VALUENUM ~ '^[0-9]+(\\.[0-9]+)?$'
""")
# 2. Tạo bảng cuối cùng, sạch sẽ bằng cách chọn từ bảng staging và chuyển đổi các cột sang các loại số chính xác của chúng.
con.execute("CREATE TABLE labevents AS SELECT SUBJECT_ID, CAST(ITEMID AS INTEGER) AS ITEMID, CAST(VALUENUM AS DOUBLE) AS VALUENUM FROM labevents_staging")
# 3. Bỏ bảng staging tạm thời để tiết kiệm không gian.
con.execute("DROP TABLE labevents_staging")
con.close()
return db_path
Thay vì cố gắng tải các tệp CSV MIMIC-III khổng lồ vào bộ nhớ bằng pandas (có khả năng sẽ bị sập), chúng ta đang sử dụng khả năng của DuckDB để xử lý dữ liệu trực tiếp từ đĩa. Việc xử lý hai giai đoạn của bảng LABEVENTS là một kỹ thuật quan trọng. Bằng cách tải dữ liệu dưới dạng văn bản trước và lọc nó trước khi chuyển sang các loại số, chúng ta xử lý các vấn đề về chất lượng dữ liệu và tạo ra một bảng cuối cùng nhỏ hơn, sạch hơn và nhanh hơn nhiều để truy vấn.
Hãy thực thi hàm để xây dựng cơ sở dữ liệu lâm sàng của chúng ta và sau đó chạy một bài kiểm tra nhanh để kiểm tra kết quả.
# Thực thi hàm để xây dựng cơ sở dữ liệu.
db_path = load_real_mimic_data()
# Nếu cơ sở dữ liệu được tạo thành công, kết nối với nó và kiểm tra schema và một số dữ liệu mẫu.
if db_path:
print(f"\nCơ sở dữ liệu MIMIC-III thực đã được tạo tại: {db_path}")
print("\nĐang kiểm tra kết nối cơ sở dữ liệu và schema...")
con = duckdb.connect(db_path)
print(f"Các bảng trong DB: {con.execute('SHOW TABLES').df()['name'].tolist()}")
print("\nMẫu của bảng 'patients':")
print(con.execute("SELECT * FROM patients LIMIT 5").df())
print("\nMẫu của bảng 'diagnoses_icd':")
print(con.execute("SELECT * FROM diagnoses_icd LIMIT 5").df())
con.close()
Đầu ra chúng ta nhận được…
OUTPUT
Attempting to load real MIMIC-III data from local CSVs...
Required files found. Proceeding with database creation.
Loading PATIENTS.csv.gz into DuckDB...
Loading DIAGNOSES_ICD.csv.gz into DuckDB...
Loading and processing LABEVENTS.csv.gz (this may take several minutes)...
Real MIMIC-III database created at: ./data/mimic_db/mimic3_real.db
Testing database connection and schema...
Tables in DB: ['patients', 'diagnoses_icd', 'labevents']
Sample of 'patients' table:
ROW_ID SUBJECT_ID GENDER DOB DOD DOD_HOSP DOD_SSN EXPIRE_FLAG
0 238 250 F 2164-12-27 2198-02-18 2198-02-18 2198-02-18 1
1 239 251 M 2078-02-21 NaN NaN NaN 0
2 240 252 M 2049-06-06 2123-09-01 2123-09-01 2123-09-01 1
3 241 253 F 2081-11-26 NaN NaN NaN 0
4 242 254 F 2028-04-12 NaN NaN NaN 0
Sample of 'diagnoses_icd' table:
ROW_ID SUBJECT_ID HADM_ID SEQ_NUM ICD9_CODE
0 129769 109 172335 1 40301
1 129770 109 172335 2 486
2 129771 109 172335 3 58281
3 129772 109 172335 4 5855
4 129773 109 172335 5 42822
Đầu ra xác nhận rằng pipeline nhập dữ liệu của chúng ta đã hoạt động. Chúng ta đã tạo thành công một cơ sở dữ liệu SQL DuckDB bền vững tại ./data/mimic_db/mimic3_real.db. Các truy vấn thử nghiệm cho thấy các bảng cốt lõi (patients, diagnoses_icd, labevents) đã được tải chính xác với các schema phù hợp.
Agent Patient Cohort Analyst của chúng ta giờ đây có quyền truy cập vào một cơ sở dữ liệu lâm sàng thực tế, hiệu suất cao chứa hàng triệu bản ghi, cho phép nó cung cấp các ước tính khả thi thực sự dựa trên dữ liệu.
 Bước tiền xử lý (Tạo bởi Fareed Khan)
Bước tiền xử lý (Tạo bởi Fareed Khan)
Cuối cùng, hãy lập chỉ mục tất cả dữ liệu văn bản phi cấu trúc của chúng ta vào các kho vector có thể tìm kiếm. Điều này sẽ làm cho các tài liệu PubMed, FDA và đạo đức có thể truy cập được bởi các agent RAG của chúng ta.
from langchain_community.document_loaders import DirectoryLoader, TextLoader
from langchain.text_splitter import RecursiveCharacterTextSplitter
from langchain_community.vectorstores import FAISS
from langchain_core.documents import Document
def create_vector_store(folder_path: str, embedding_model, store_name: str):
"""Tải tất cả các tệp .txt từ một thư mục, chia chúng thành các đoạn và tạo một kho vector FAISS trong bộ nhớ."""
print(f"--- Đang tạo Kho Vector {store_name} ---")
# Sử dụng DirectoryLoader để tải hiệu quả tất cả các tệp .txt từ thư mục được chỉ định.
loader = DirectoryLoader(folder_path, glob="**/*.txt", loader_cls=TextLoader, show_progress=True)
documents = loader.load()
if not documents:
print(f"Không tìm thấy tài liệu nào trong {folder_path}, bỏ qua việc tạo kho vector.")
return None
# Sử dụng RecursiveCharacterTextSplitter để chia các tài liệu lớn thành các đoạn nhỏ hơn, 1000 ký tự với 100 ký tự chồng chéo.
# Sự chồng chéo giúp duy trì ngữ cảnh giữa các đoạn.
text_splitter = RecursiveCharacterTextSplitter(chunk_size=1000, chunk_overlap=100)
texts = text_splitter.split_documents(documents)
print(f"Đã tải {len(documents)} tài liệu, được chia thành {len(texts)} đoạn.")
print("Đang tạo embeddings và lập chỉ mục vào FAISS... (Quá trình này có thể mất một chút thời gian)")
# FAISS.from_documents là một hàm tiện lợi xử lý cả việc embedding các đoạn văn bản
# và xây dựng chỉ mục FAISS hiệu quả trong một bước.
db = FAISS.from_documents(texts, embedding_model)
print(f"Kho Vector {store_name} đã được tạo thành công.")
return db
def create_retrievers(embedding_model):
"""Tạo các trình truy xuất kho vector cho tất cả các nguồn dữ liệu phi cấu trúc và hợp nhất tất cả các kho tri thức."""
# Tạo một kho vector chuyên biệt, riêng biệt cho mỗi loại tài liệu.
pubmed_db = create_vector_store(data_paths["pubmed"], embedding_model, "PubMed")
fda_db = create_vector_store(data_paths["fda"], embedding_model, "FDA")
ethics_db = create_vector_store(data_paths["ethics"], embedding_model, "Ethics")
# Trả về một từ điển duy nhất chứa tất cả các công cụ truy cập dữ liệu đã được cấu hình.
# Phương thức 'as_retriever' chuyển đổi kho vector thành một đối tượng Retriever LangChain tiêu chuẩn.
# Tham số 'k' trong 'search_kwargs' kiểm soát số lượng tài liệu hàng đầu được trả về bởi một tìm kiếm.
return {
"pubmed_retriever": pubmed_db.as_retriever(search_kwargs={"k": 3}) if pubmed_db else None,
"fda_retriever": fda_db.as_retriever(search_kwargs={"k": 3}) if fda_db else None,
"ethics_retriever": ethics_db.as_retriever(search_kwargs={"k": 2}) if ethics_db else None,
"mimic_db_path": db_path # Chúng ta cũng bao gồm đường dẫn tệp đến cơ sở dữ liệu DuckDB có cấu trúc của mình.
}
Hàm create_vector_store này là cách tiếp cận để tạo các cơ sở tri thức sẵn sàng cho RAG từ các tệp văn bản. Nó đóng gói mẫu "tải -> chia -> nhúng -> lập chỉ mục" phổ biến. Hàm create_retrievers sau đó điều phối quá trình này, tạo ra một kho vector riêng biệt, chuyên biệt cho mỗi loại tài liệu của chúng ta.
Thay vì một kho vector khổng lồ duy nhất, chúng ta có các kho nhỏ hơn, chuyên biệt theo lĩnh vực. Điều này cho phép các agent của chúng ta thực hiện các tìm kiếm có mục tiêu và hiệu quả hơn (ví dụ: Regulatory Specialist sẽ chỉ truy vấn fda_retriever).
Hãy chạy hàm cuối cùng để xây dựng bộ kho tri thức hoàn chỉnh của chúng ta.
# Thực thi hàm để tạo tất cả các trình truy xuất của chúng ta.
knowledge_stores = create_retrievers(llm_config["embedding_model"])
print("\nCác kho tri thức và trình truy xuất đã được tạo thành công.")
# In từ điển cuối cùng để xác nhận tất cả các thành phần đều có mặt.
for name, store in knowledge_stores.items():
print(f"{name}: {store}")
OUTPUT
--- Creating PubMed Vector Store ---
100%|██████████| 20/20 [00:00<00:00, 1102.77it/s]
Loaded 20 documents, split into 35 chunks.
Generating embeddings and indexing into FAISS... (This may take a moment)
Batches: 100%|██████████| 2/2 [00:03<00:00, 1.70s/it]
PubMed Vector Store created successfully.
--- Creating FDA Vector Store ---
100%|██████████| 1/1 [00:00<00:00, 137.95it/s]
Loaded 1 documents, split into 48 chunks.
Generating embeddings and indexing into FAISS... (This may take a moment)
Batches: 100%|██████████| 2/2 [00:04<00:00, 2.08s/it]
FDA Vector Store created successfully.
--- Creating Ethics Vector Store ---
100%|██████████| 1/1 [00:00<00:00, 143.20it/s]
Loaded 1 documents, split into 1 chunks.
Generating embeddings and indexing into FAISS... (This may take a moment)
Batches: 100%|██████████| 1/1 [00:00<00:00, 2.62it/s]
Ethics Vector Store created successfully.
Knowledge stores and retrievers created successfully.
pubmed_retriever: VectorStoreRetriever(tags=['FAISS', 'OllamaEmbeddings'], vectorstore=<...>)
fda_retriever: VectorStoreRetriever(tags=['FAISS', 'OllamaEmbeddings'], vectorstore=<...>)
ethics_retriever: VectorStoreRetriever(tags=['FAISS', 'OllamaEmbeddings'], vectorstore=<...>)
mimic_db_path: ./data/mimic_db/mimic3_real.db
Đầu ra xác nhận rằng toàn bộ tri thức của chúng ta hiện đã được lắp ráp và vận hành đầy đủ. Chúng ta đã xử lý thành công tất cả các nguồn văn bản phi cấu trúc của mình, PubMed, FDA và Ethics, thành các kho vector FAISS có thể tìm kiếm.
Từ điển knowledge_stores cuối cùng là kho lưu trữ công cụ truy cập dữ liệu hoàn chỉnh, tập trung của chúng ta. Nó chứa mọi thứ mà guild agent của chúng ta sẽ cần để thực hiện nghiên cứu của mình.
Với dữ liệu đã được tải xuống, xử lý và lập chỉ mục, và các LLM của chúng ta đã được cấu hình, bây giờ chúng ta có thể bắt đầu xây dựng thành phần chính đầu tiên của hệ thống agent của mình: Guild Thiết kế Thử nghiệm.
Xây dựng mạng lưới thiết kế thử nghiệm nội bộ
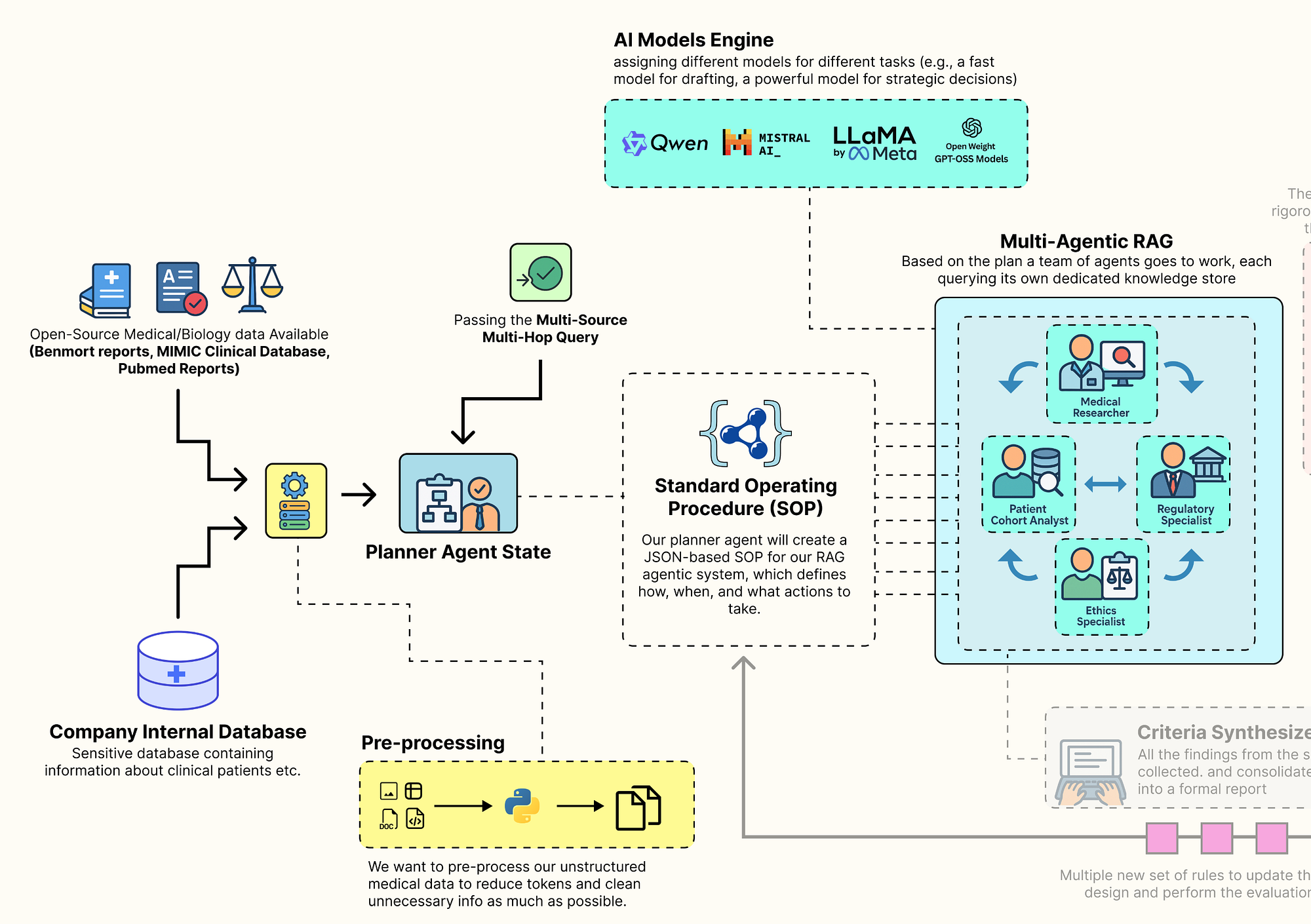
Với cơ sở tri thức của chúng ta đã sẵn sàng, bây giờ chúng ta có thể xây dựng phần cốt lõi của hệ thống. Đây sẽ không phải là một chuỗi RAG tuyến tính, đơn giản. Nó là một luồng công việc cộng tác, đa agent được xây dựng bằng LangGraph, nơi một nhóm các chuyên gia AI làm việc cùng nhau để biến một khái niệm thử nghiệm cấp cao thành một tài liệu tiêu chí chi tiết, dựa trên dữ liệu.
 RAG vòng lặp nội bộ chính (Tạo bởi Fareed Khan)
RAG vòng lặp nội bộ chính (Tạo bởi Fareed Khan)
Hành vi của toàn bộ kiến trúc này không được mã hóa cứng. Thay vào đó, nó được điều chỉnh bởi một đối tượng cấu hình động duy nhất mà chúng ta gọi là Quy trình Vận hành Tiêu chuẩn (GuildSOP).
SOP này là "bộ gen" của pipeline RAG của chúng ta, và chính bộ gen này mà "Giám đốc Nghiên cứu AI" vòng lặp ngoài của chúng ta sẽ học cách phát triển và tối ưu hóa.
Trong phần này, đây là những gì chúng ta sẽ làm:
- Định nghĩa bộ gen RAG: Chúng ta sẽ tạo mô hình Pydantic
GuildSOP, một cấu hình có cấu trúc sẽ kiểm soát mọi khía cạnh của luồng công việc kiến trúc thẻ. - Kiến trúc không gian làm việc chung: Chúng ta sẽ định nghĩa
GuildState, không gian trung tâm nơi các agent của chúng ta sẽ chia sẻ kế hoạch và phát hiện của họ. - Xây dựng các agent chuyên biệt: Chúng ta sẽ triển khai mỗi chuyên gia, Planner, Researchers, SQL Analyst, và Synthesizer như một hàm Python riêng biệt sẽ đóng vai trò là một nút trong đồ thị của chúng ta.
- Điều phối sự cộng tác: Chúng ta sẽ kết nối các nút agent này lại với nhau bằng
LangGraphđể định nghĩa luồng công việc hoàn chỉnh, từ đầu đến cuối của Guild. - Thực hiện một lần chạy thử đầy đủ: Chúng ta cũng sẽ gọi toàn bộ đồ thị Guild đã biên dịch với SOP cơ sở của mình để xem nó hoạt động và tạo ra tài liệu tiêu chí đầu tiên của chúng ta.
Định nghĩa SOP của Guild
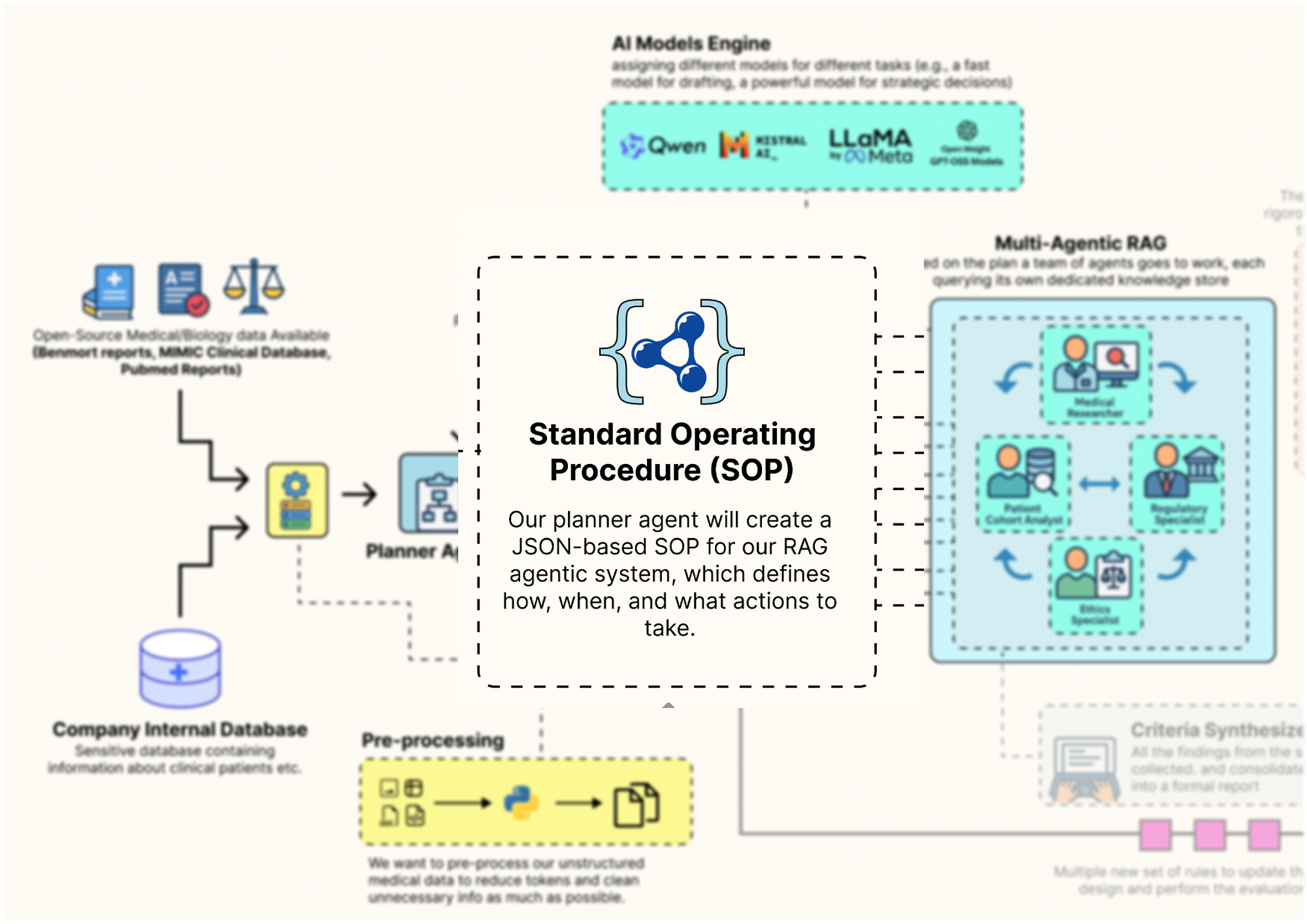
Đầu tiên, chúng ta cần định nghĩa cấu trúc sẽ kiểm soát toàn bộ luồng hành vi. Chúng ta sẽ sử dụng một BaseModel của Pydantic để tạo GuildSOP của mình. Đây là một lựa chọn thiết kế quan trọng. Sử dụng Pydantic cho chúng ta một đối tượng cấu hình được định kiểu, xác thực và tự tài liệu hóa.
 Thiết kế SOP của Guild (Tạo bởi Fareed Khan)
Thiết kế SOP của Guild (Tạo bởi Fareed Khan)
GuildSOP này là phần trung tâm mà Giám đốc AI vòng lặp ngoài của chúng ta sau này sẽ biến đổi và phát triển, vì vậy việc có một schema nghiêm ngặt là quan trọng cho một quá trình tiến hóa ổn định. Hãy lập trình điều đó.
from pydantic import BaseModel, Field
from typing import Literal
class GuildSOP(BaseModel):
"""Quy trình Vận hành Tiêu chuẩn cho Guild Thiết kế Thử nghiệm. Đối tượng này hoạt động như cấu hình động cho toàn bộ luồng công việc RAG."""
# Trường này chứa prompt hệ thống cho Agent Planner, quyết định chiến lược của nó.
planner_prompt: str = Field(description="Prompt hệ thống cho Agent Planner.")
# Tham số này kiểm soát số lượng tài liệu mà Medical Researcher truy xuất, cho phép chúng ta tinh chỉnh độ rộng của tìm kiếm.
researcher_retriever_k: int = Field(description="Số lượng tài liệu để Medical Researcher truy xuất.", default=3)
# Đây là prompt hệ thống cho người viết cuối cùng, Agent Synthesizer.
synthesizer_prompt: str = Field(description="Prompt hệ thống cho Agent Tổng hợp Tiêu chí.")
# Điều này cho phép chúng ta thay đổi động mô hình được sử dụng cho giai đoạn soạn thảo cuối cùng, đánh đổi giữa tốc độ và chất lượng.
synthesizer_model: Literal["qwen2:7b", "llama3.1:8b-instruct"] = Field(description="LLM sẽ sử dụng cho Synthesizer.", default="qwen2:7b")
# Các biến boolean này hoạt động như "cờ tính năng," cho phép Giám đốc bật hoặc tắt toàn bộ khả năng của agent.
use_sql_analyst: bool = Field(description="Có sử dụng agent Patient Cohort Analyst hay không.", default=True)
use_ethics_specialist: bool = Field(description="Có sử dụng agent Ethics Specialist hay không.", default=True)
Lớp GuildSOP không chỉ là một tệp cấu hình, nó là một tài liệu sống định nghĩa chiến lược hiện tại của Guild. Bằng cách để lộ các tham số chính như prompt, cài đặt trình truy xuất (researcher_retriever_k), và thậm chí cả việc sử dụng agent nào (use_sql_analyst), chúng ta đang tạo ra một bộ chiến lược mà Giám đốc AI vòng lặp ngoài của chúng ta có thể kéo để tinh chỉnh toàn bộ hiệu suất.
Chúng ta đang sử dụng Literal cho synthesizer_model để đảm bảo an toàn kiểu dữ liệu để Giám đốc chỉ có thể chọn từ một danh sách các mô hình hợp lệ đã được định nghĩa trước.
Bây giờ chúng ta đã có bản thiết kế cho SOP của mình, hãy tạo một instance cụ thể, phiên bản 1.0. baseline_sop này sẽ là điểm khởi đầu của chúng ta, chiến lược ban đầu, được thiết kế thủ công mà chúng ta sẽ giao cho Giám đốc AI của mình cải thiện.
import json
# Chúng ta khởi tạo lớp GuildSOP của mình với một bộ giá trị cơ sở, mặc định.
baseline_sop = GuildSOP(
# Prompt ban đầu của planner rất chi tiết, hướng dẫn agent về vai trò của nó, các chuyên gia có sẵn, và định dạng đầu ra JSON bắt buộc.
planner_prompt="""Bạn là
Theo dõi trên X